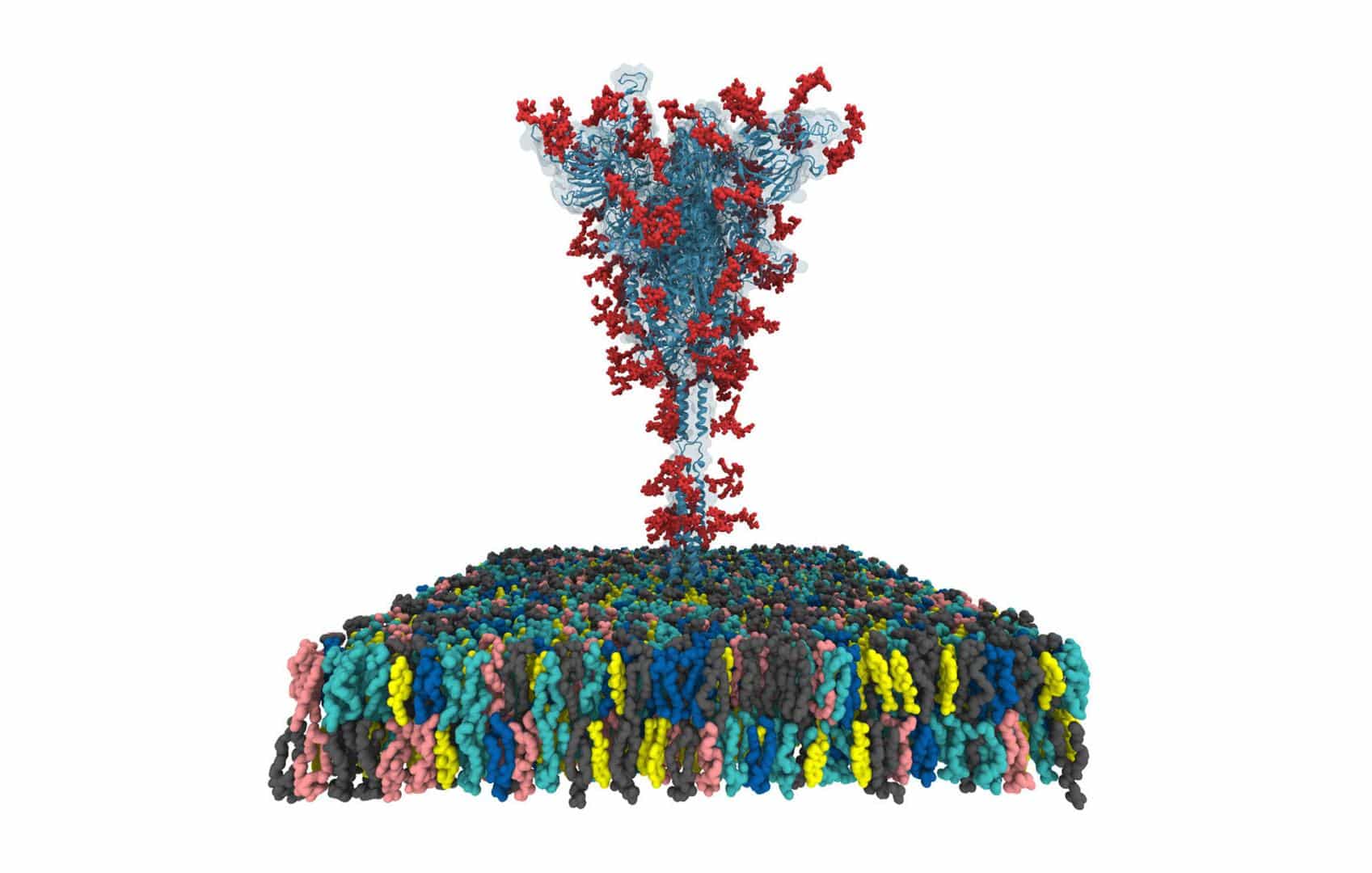
O Sars-Cov-2, vírus causador da Covid-19, utiliza “espinhos” de proteína S para romper a proteção das células pulmonares. Eles cobrem toda superfície do vírus como “joias numa coroa” – daí que veio o nome “coronavírus”. Mas estas estruturas, que tornam o vírus tão perigoso, podem ser também seu ponto fraco.
Um grupo de pesquisadores da Universidade Nacional de Seul, na Coreia do Sul, da Universidade de Cambridge, no Reino Unido, e da Universidade de Lehigh, nos EUA, fizeram um mapeamento completo de todos os átomos de uma proteína S. Esse modelo, detalhado em um estudo publicado no Journal of Physical Chemistry B, pode ser usado para o desenvolvimento de vacinas e medicamentos antivirais.
O programa usado na criação do modelo, o CHARMM-GUI, é de acesso público e pode ser encontrado na página do projeto. O software simula sistemas biomoleculares complexos de maneira simples, e é descrito como um “microscópio computacional” que permite aos cientistas entender interações em nível molecular que não podem ser observadas de outra maneira.
“São os primeiros modelos de proteína ‘spike’ (S) do Sars-Cov-2 totalmente glicosilados que estão disponíveis para outros cientistas”, afirma o professor do Departamento de Ciências Biológicas e Bioengenharia da Universidade de Lehigh, Wonpil Im, criador do CHARMM-GUI.
Além das proteínas S do coronavírus, os pesquisadores modelaram todos os potenciais carboidratos que se ligam aos espinhos e impedem o reconhecimento de anticorpos, o que dificulta o desenvolvimento de uma vacina.